 |
| トップページ > リボソームによるタンパク質合成の1分子力学測定 |
 |
||||||||||||||||||||||||||
リボソームによるタンパク質合成の1分子力学測定
|
||||||||||||||||||||||||||
 |
上村 想太郎 (Sotaro Uemura) 東京大学大学院薬学系研究科 |
[Summary]
The ribosome is a molecular machine that translates the genetic code contained on the messenger RNA (mRNA) into an amino acid sequence through repetitive cycles of transfer RNA (tRNA) selection, peptide bond formation and translocation. Here we demonstrate an optical tweezer assay to measure the rupture force between a single ribosome complex and mRNA. The rupture force was compared between ribosome complexes assembled on an mRNA with and without a strong Shine-Dalgarno (SD) sequence. The removal of the SD sequence significantly reduced the rupture force, indicating that the SD interactions contribute significantly to the stability of the ribosomal complex on the mRNA in a pre-peptidyl transfer state. In contrast, the post-peptidyl transfer state weakened the rupture force as compared to the complex in a pre-peptidyl transfer state and it was the same for both the SD-containing and SD-deficient mRNAs. The results suggest that formation of the first peptide bond destabilizes the SD interaction, resulting in the weakening of the force with which the ribosome grips an mRNA. This might be an important requirement to facilitate movement of the ribosome along mRNA during the first translocation step.
キーワード:1分子計測、リボソーム、光ピンセット法、破断力、Shine-Dalgarno配列(SD配列)、アロステリック効果
1.はじめに
|
30Sと50Sサブユニットで構成される細菌リボソームはその3分の1がタンパク質であり、残りの3分の2はリボソームRNA(rRNA)で出来ている。リボソームはアミノ酸配列によってコードされたmRNA上のコドンに対応したアミノアシルtRNAを結合し続け、それによって毎秒10〜20のアミノ酸を合成することが できるが、その正確性はかなり高く、誤りは約3000アミノ酸に1回の確率である1)。複雑な構造を持つリボソーム分子はどのようにしてこのような高い正確性を保ちながらmRNA上のコドンを読み取り、正確にタンパク質を合成できるのだろうか?この生命現象の重要な謎は長年にわたる生化学的手法などを用いた研究によってその巧妙な仕組みが徐々に明らかにされてきた。さらに構造解析による手法の発展によって近年高分解能で構造情報が明らかとなり、より詳細な構造機能が議論されるようになった(Fig.1)2)-5)。 |
|
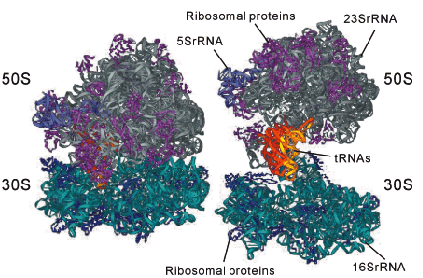 |
Figure 1 The structure of the complete Thermus thermophilus 70S ribosome was crystallized in a complex including the 30S subunit (16S rRNA and small subunit proteins), 50S subunit (23S rRNA, 5S rRNA, and large subunit proteins), P- and E-site tRNA, and solved by x-ray crystallography to a resolution of 5.5 Å2). |
リボソーム機能の大きな特徴の1つとして50Sサブユニットの大部分を占めている23SrRNAにはペプチジルトランスフェラーゼと呼ばれるペプチド結合を触媒するRNA酵素(リボザイム)としての機能を持っていることが挙げられる。一方、30Sサブユニットの16SrRNAはmRNAと相互作用し、mRNA上のコドンとtRNA上のアンチコドン結合を保護するような構造配置をする。それぞれの機能を阻害するような抗生物質は翻訳活性を著しく低下あるいは停止させることが知られている。mRNA上の遺伝情報を正確にタンパク質へと翻訳するためにはこれらの重要な2つの機能が独立に働くのではなく、互いに協同的に働かなければならず、そこには30S-50S間に機能する巧妙な共役メカニズムが存在するはずである。しかし、これらの現象を明らかにするためには先に述べた従来までの生化学的手法や構造解析によるアプローチでは限界がある。生化学的手法を用いた多分子系によるアプローチではデータが平均化されてしまうために個々の分子の特性を直接捉えることは難しい。また、構造学的手法では試料を結晶化させることがほとんどのため、相互作用が時間的にどのように変化するのかを捉えるのは非常に難しい。
そこで著者らはタンパク質翻訳系を担うリボソームの翻訳初期複合体に対して光ピンセット法を用いた1分子力測定を応用し、mRNAと30Sサブユニットとの間に働く相互作用の力を各翻訳過程に対して測定することでサブユニット間に働く共役現象を直接1分子レベルで明らかにした。その結果、初期結合配列であるSD(Shine-Dalgarno)配列部位の結合が、50S内のペプチド結合によって弱められていることがわかった。この結果は30S-50Sのサブユニット間にアロステリック相互作用があることを示しており、このことがmRNA上を移動する分子モーターとしての機能として重要なメカニズムであると考えている6)。本文では1分子力測定を用いたこのような著者の結果を光ピンセット法の手法と共に示し、今後の応用展開について展望する。
2.リボソーム翻訳サイクル
リボソーム研究の初期、ペプチド結合を形成するためには最低2つのアミノアシルtRNAが必要であると考えられたため、リボソームにはアミノアシルtRNA(A部位)とぺプチジルtRNA(P部位)の2つのtRNAが結合できるための結合部位は2つしか存在しないと考えられていたが、Exit部位(E部位)の発見によっ て現在のスリーサイトモデルが確立されるようになった(Fig.2)7,8)。リボソームが連続的にペプチド結合を合成し続けるためにはGTP加水分解酵素であるEF-Tuと伸長因子であるEF-Gが必要不可欠である。EF-TuはGTP加水分解作用によりアミノアシルtRNAをA部位に運ぶ役割があり、EF-GはGTP加水分解作用によってA部位、P部位にある2つのtRNAをそれぞれP部位とE部位にトランスロケーションすることが知られている。その結果、リボソームはmRNAに対して1コドン分だけ3'方向へ移動することになる。このトランスロケーションによってA部位に結合していたtRNAがP部位へと移動するためにA部位に再び新しいtRNAがEF-Tuによって運ばれるようになる。
|
3.光ピンセット法
近年,蛍光顕微鏡技術の進歩に伴い光ピンセット法が研究の進展に大きく寄与している。光ピンセット法とはAshkinらが開発した手法9)で、大きな開口数(N.A.)の対物レンズを用いてレーザー光を集光することにより、直径が数十nmから数μmのビーズ粒子をその焦点付近に捕捉し、自由自在に操作する技術である。ビーズには捕捉中心からの変位に比例した復元力が働くことから、顕微画像解析法や四分割フォトダイオードを用いて微粒子の変位をnm精度で計測すると、ビーズに加わる力をpNの精度で計測できる。具体的には、キネシンやミオシンなどに代表されるようなATP加水分解によって生じる化学エネルギーを運動エネルギーに変換する分子モーター1分子をビーズに結合し、分子モーターの動きや発生力をビーズの動きを通して計測する。ビーズを捕捉することで、間接的だが分子モーターの変位を精度よく計測することができる。ビーズを使うもう一つの利点は、分子のブラウン運動(熱運動)を抑えることにある。光ピンセット法で用いるレーザー光の波長は通常近赤外光である(YAGレーザーによる1064nmなど)。その理由として蛍光標識観察の邪魔になる可視領域を避けること、生体物質にダメージを与える紫外領域から遠いこと、また数μmの赤外領域になると水分子による吸収があり温度上昇があるのでこれを避けることなどが挙げられる。
4.光ピンセット法によるリボソーム1分子破断力測定
翻訳初期過程ではリボソームはmRNA初期コドンのAUGと数塩基ほど離れた位置に存在するShine-Dalgarno(SD)配列と呼ばれるプリンに富んだ配列(AGGA)を30S内の16SrRNAの3'末端部分と相補的に結合させることが知られている10,11)。しかし、翻訳が開始されるとリボソームはmRNA上を移動しなければならないため、いずれはこのSD配列における翻訳初期結合を解除しなければ移動することができないはずである。ではいつどのようにリボソームはこの結合を解除し、mRNA上の移動を可能にしているのだろうか?。そこには翻訳初期結合を解除するシグナルが存在し、タンパク質合成と関係があると予想することができる。
そこで我々は光ピンセット法を用いて1分子のリボソーム翻訳初期複合体における30SとmRNAに働く相互作用の力をペプチド結合前後で測定した。
まず、リボソームはSD配列を含んだT4gene32由来の57塩基からなるmRNAと複合体を形成させた。mRNAは5'末端がビオチン化されており、活性を保ったままガラス面上に複合体を固定させることができる。用いたリボソームの30S内16S rRNAのへリックス44部位には23塩基の付加配列が遺伝子工学的に導入されており12)相補的な配列を持ち、3'末端にDigoxigenin抗体が付加されたオリゴDNAを用いることでリボソーム30Sをビーズ粒子と結合させた。ビーズ粒子は直径1μmのCarboxylateビーズを用いており、EDC(Ethyl Dimethylaminopropyl Carbodiimide)によってAnti-digoxigenin抗体と架橋され、相補オリゴを介して直接結合させた(Fig.3a,b)。1分子のリボソーム複合体が1つのビーズ粒子と結合しているかどうかはCy3蛍光色素でラベルしたtransfer RNA(tRNA)を結合した複合体を用いてその蛍光シグナルが1段階で退色する過程を観察することで確認することができた。
(a)The molecular attachments within the mRNA-ribosome-bead complex. Ribosomal particles were assembled on a short mRNA tethered to the surface via biotin-streptavidin linkage. A digoxigenin-modified oligonucleotide was designed to hybridize to an rRNA loop extension on the small ribosomal subunit. A bead coated with anti-digoxigenin antibody was conjugated to the oligonucleotide and used for optical trapping of the ribosomal complex.(b)The tethered ribosome-bead complex fluctuates around the point of surface attachment.(c)Optical tweezers are used to trap the bead.(d)As the stage with the attached ribosome-bead complex is moved in one direction,the force exerted on the complex increases and the bead becomes displaced. (e)Eventually the external force becomes sufficient to rupture the complex,and the bead returns to the trap center position. |
次にこの1分子力測定系を用いて破断力測定を試みた。まず、mRNAを介してガラス面上に固定されたビーズ複合体を光ピンセットで光捕捉し(Fig.3d)。するとビーズ複合体に負荷が徐々にかかり最終的に30SとmRNAの結合が破断し、ビーズが捕捉中心へとすばやく引き戻される(Fig.3e)。負荷をかけ始めてから破断現象が起こるまでの時間はリボソームのmRNAからの解離時間に比べて十分速いものであるため、安定した複合体に対して測定を行っている。
Fig.4aに破断力測定の例をいくつか示した。破断現象はmRNAからのリボソームの解離の結果起こるものであると考えられる。これを証明するために、以下の予備実験を行った。はじめに、リボソームをmRNA非存在下でガラス表面に直接共有結合させ、破断力測定を試した(Fig.4b)。さらに30Sに遺伝子工学的に導入された付加配列と同配列をもつオリゴDNAをビオチン・アビジン結合を介してガラス面上に固定し、同様の方法でビーズ粒子と結合させ、破断力測定を試みた(Fig.4c)。しかし、どちらの場合も結合力は強力だったので光ピンセットで破断できる範囲を超えていた。これらの結果は、rRNAに導入したオリゴDNAと相補的なオリゴとの結合が十分強いことを意味している。
|
もうひとつの予備実験として、破断測定後にオリゴ結合が破断していないかどうかを確認するために今回のみテトラメチルローダミン蛍光色素でラベルしたtRNAを複合体に結合させ、破断力測定後に蛍光シグナルの確認を行った。その結果、破断測定後蛍光シグナルが破断後のビーズ表面上に確認されたため、複合体がmRNAから破断していると考えることができる。リボソームとビーズの結合がこの測定条件下において十分強いことが改めて明らかとなった。テトラメチルローダミン蛍光色素を用いた理由は光ピンセットで用いた1064nmの赤外レーザーによる蛍光色素の退色の影響が比較的少ない色素であるからである。
5.破断力分布によるペプチド結合前後の力変化と初期配列結合の影響
破断現象とはポテンシャル障壁を外力による仕事の分だけ下げて結合安定から障壁を越えると起こる。つまり、どれだけの仕事を外から加えると障壁を越えることができるのかを調べることができる。各翻訳過程では結合安定が異なると考えられるので、30SとmRNAの結合には各過程でどのような状態が存在するのかを調べることができる。注意すべき点は、この現象が確率的であるために繰り返し測定をして、統計的に議論しなければならないことである。まず、tRNA非存在下においてリボソームとmRNAの結合を調べた。リボソームはSD配列部分でmRNAと結合しているため、tRNA非存在下でも複合体は安定である。この条件で繰り返し測定を行った結果、破断力は10.6 pNをピークにもつ分布を示した(Fig.5a)。さらに脱アミノアシル化tRNAfMetをP部位に結合した複合体は5pNほど強いピークを持つ破断力分布となった(Fig.5b)。続けてアミノアシル化tRNAであるPhe-tRNAPheをA部位へと結合させるとさらに10 pNほど強い破断力(26.5pN)を示した(Fig.5c)。
All complexes were assembled in 5 mM Mg2+. (a, f) Ribosome-mRNA complex without tRNAs. No tethered beads were observed in the absence of the SD sequence(f),indicating that the complex was too unstable for a force measurement under these conditions.(b,g)Ribosome-mRNA complex carrying deacylated tRNAfMet in the P site.(c, h)Ribosome-mRNA complex with tRNAfMet in the P site and Phe-tRNAPhe in the A site. (d,i) Ribosome-mRNA complex with tRNAfMet in the P site and N-acetyl-Phe-tRNAPhe in the A site.(e) Ribosome-mRNA complex after ribosome-catalyzed peptide bond formation. The complex was assembled with fMet-tRNAfMet in the P site and Phe-tRNAPhe in the A site, and incubated for 20 min to allow for peptidyl transfer. |
ここでtRNAがリボソームから解離した複合体に対して破断力測定を行っている可能性を否定するために、tRNA存在下の全ての実験条件においてtRNAfMetはtRNAの8番目のUをCy3-maleimideでラベルし、tRNAPheは47番目のUをCy5-NHSでラベルしたtRNAを用いた13)。破断力測定直前に532nmレーザーによる励起によってその蛍光シグナルを確認することでその複合体からtRNAが解離していないことを確認した。A部位及びP部位にそれぞれtRNAが結合している場合はCy3とCy5の間でFRET (Fluorescence Resonance Energy Transfer)が観測されることを利用して、FRETシグナルを検出した複合体のみを 破断力測定に用いた(Fig. 6)。
|
ここまでの結果から、P部位とA部位に同時にtRNAが結合するとリボソーム複合体はさらにmRNAとの相互作用が強くなり、安定化することがわかった。次にペプチドtRNAのアナログであるN-acetyl-Phe-tRNAPheをA部位に結合した複合体に対して破断力測定を行うと、驚くべきことに12.7pNとなり、Phe-tRNAPheをA部位に結合した複合体の場合(26.5pN)と比べて著しく低い破断力を示した(Fig.5d)。このことはペプチド結合を形成するとリボソームとmRNAの相互作用が弱くなることを示唆している。これをさらに確かめるためにfMet-tRNAfMetをP部位、Phe-tRNAPheをA部位に結合させ、リボソーム自身の触媒作用によるfMetとPheのペプチド結合形成を行い、同様に破断力を測定すると、11.4 pNのピークを持つ値が現れただけでなく24.8pNの大きな値も少数ではあるが計測された(Fig.5e)。この少数の大きな値(14.3%)は触媒活性の不活性が原因であると思われる。よってこの結果からペプチド結合形成がリボソームとmRNAの相互作用を不安定化していることが明らかとなった。
ここで、リボソームとmRNAの相互作用のどの部位が不安定化しているのかを突き止めるためにSD初期結合に着目した。複合体の安定化にSD相互作用がどのように影響しているのかを調べるために、mRNAのSD配列部分をAGGAからACCAへと変化させ、SD部分で相互作用しないmRNA14)を用いて破断力測定を同様に行った。tRNA非存在下ではSD相互作用できないため、複合体は不安定となり、測定可能なビーズ複合体は観測されなかった(Fig.5f)。一方、tRNAfMetをP部位へ結合した複合体は測定が可能となり、5pNほどの小さな破断力が測定された(Fig.5g)。さらにPhe-tRNAPheをA部位に結合させる条件で測定すると、Fig.5cの場合と同様に10pNほど強いピーク(14.8pN)を持つ破断力分布となった(Fig.5h)。そしてN-acetyl-Phe-tRNAPheをA部位に結合した複合体で測定すると、12.1pNにピークを持つ破断力が測定された(Fig.5i)。この値はFig.5hに比べるとやや小さい値であるが、SD配列を含んだmRNAを用いた場合(Fig.5d,12.7pN)とほとんど変わらない値を示した。すなわち、SD配列を含んでいるmRNAを用いた場合とSD配列を含んでいないmRNAを用いた場合で値が変化しないという結果はペプチド結合形成によってSD相互作用が不安定化しているということを示していることに他ならない。ペプチド結合の形成はリボソーム50S内23SrRNAのペプチジルトランスフェラーゼによって行われており、SD結合はリボソーム30S内16SrRNAのアンチSD配列とmRNA上のSD配列との相互作用である。つまりこの結果は50S内のペプチド結合形成シグナルが何らかの構造変化を起こし、50S-30Sの結合面を介して30SのSD相互作用を不安定にし、トランスロケーションによるmRNA上の移動をより容易にしているのではないかと考えられる。このようなアロステリック現象は翻訳初期段階においてリボソームがmRNA上を運動する上でとても重要な機能であると考えられる。
6.今後の展望
以上のように翻訳初期複合体に対して1分子力学測定を応用した結果、新しい機能が明らかとなった。しかし、より詳しいリボソーム内部の構造変化を明らかにするためには負荷を加えた際の内部構造を同時に測定する技術が必要であろう。そのためには内部構造変化を捉えるためのFRET法を1分子力学測定と組み合わせて同時測定系を構築することが求められる。さらには翻訳機能を可視化することも必要であろう。こうした技術の発展の先には翻訳機能を光ピンセットによる力で操作できるようになることが予想され、翻訳機能に関わる病気や抗生物質による翻訳阻害機構などが1分子レベルで理解されることが期待される。
参考文献
1) F. Bouadloun, D. Donner,and C. G. Kurland, EMBO. J.,2,1351 (1983).
2) M. M. Yusupov,G. Z. Yusupova, A. Baucom,K. Lieberman,T. N. Earnest,J. H. Cate,and H. F. Noller,Science,292, 883(2001).
3) G. Z. Yusupova,M. M. Yusupov,J. H. Cate,and H. F. Noller,Cell, 106,233(2001).
4) D. Moazed,and H. F. Noller,Cell, 57,585 (1989).
5) D. Moazed, and H. F. Noller,J. Mol. Biol.,211,135(1990).
6) S. Uemura, M. Dorywalska,T. H. Lee,H. D. Kim,J. D. Puglisi,and S. Chu,Nature,446,454 (2007).
7) H. J. Rheinberger,H. Sternbach,and K. H. Nierhaus,Proc. Natl. Acad. Sci. U.S.A.,78,5310(1981).
8) M. Valle,J. Sengupta,N. K. Swami,R. A. Grassucci,N. Burkhardt,K. H. Nierhaus,R. K. Agrawal,and J. Frank,EMBO. J.,21,3557(2002).
9) A. Ashkin,J. M. Dziedzic,J. E. Bjorkholm,and S. Chu,Optics Letters.,11,288(1986).
10) J. Shine,and L. Dalgarno,Proc. Natl. Acad. Sci. U.S.A.,71, 1342(1974).
11) R. A. Calogero,C. L. Pon,M. A. Canonaco,and C. O. Gualerzi,Proc. Natl. Acad. Sci. U.S.A.,85,6427(1988).
12) M. Dorywalska, S. C. Blanchard, R. L. Gonzalez, H. D. Kim, S. Chu,and J. D. Puglisi,Nucl. Acids Res.,33, 182 (2005).
13) B. S. Watson,T. L. Hazlett,J. F. Eccleston,C. Davis,D. M. Jameson,and A. E. Johnson,Biochemistry,34,7904 (1995).
14) K. Fredrick,and H. F. Noller,Mol. Cell.,9,1125(2002).
| 氏名 | 上村想太郎(Sotaro Uemura) |
| 年齢 | 29歳 |
| 所属 | 東京大学大学院薬学系研究科 助教 |
| 所在地 | 〒133-0033東京都文京区本郷7-3-1 東京大学薬学系総合研究棟6階 TEL:03-5841-4761 FAX:03-5802-3339 E-mail:uemura@mol.f.u-tokyo.ac.jp |
| 出身大学 | 早稲田大学大学院理工学研究科 |
| 学位 | 博士(理学) |
| 現在の研究テーマ | リボソームにおけるタンパク質翻訳のメカニズムの研究 |
| Copyright(c) 1996-2007 DOJINDO LABORATORIES,ALL Rights Reserved. |
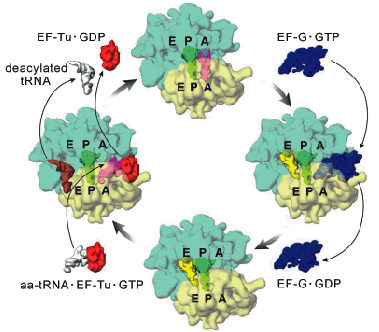 Figure 2
Figure 2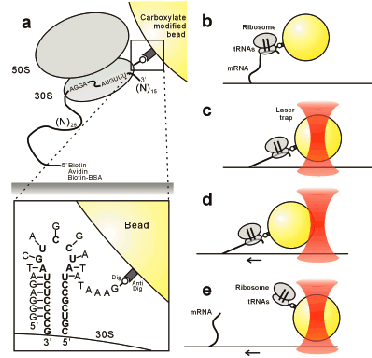

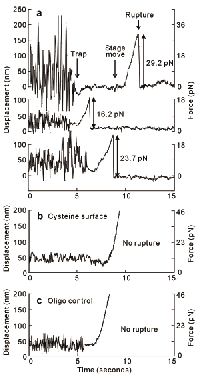
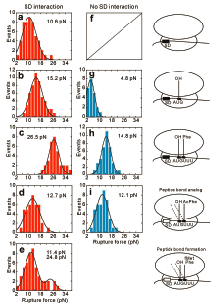
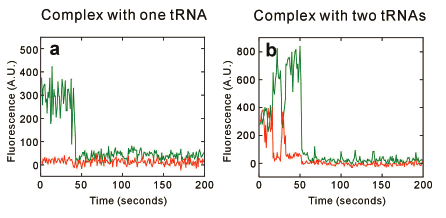 Figure 6
Figure 6