小社が立地する熊本県の生命科学研究最前線を、熊本大学の若手研究者が連載(8回)でお届けします。
栄養素代謝物によるエピゲノム制御の分子メカニズム
Nutritional control of the epigenome

日野 信次朗
熊本大学
発生医学研究所
細胞医学分野
准教授
Abstract Organisms fine-tune their gene expression in response to environmental cues such as nutrients, oxygen, and pathogens, thereby enabling environmental adaptation. Epigenetic gene regulation, through DNA methylation and histone modifications, is essential for this adaptive gene expression. In particular, food-derived metabolites such as acetyl-CoA, S-adenosylmethionine, and flavin adenine dinucleotide play central roles, as they serve either as substrates for the modifications or as essential cofactors for the modifying enzymes. This review will focus on the specific mechanisms by which metabolites regulate DNA/histone modifications, especially in light of newly identified metabolite-epigenome relationships.
1. はじめに
環境に応じた適切な遺伝子発現において、DNA やヒストンタンパク質の化学修飾であるエピゲノムが重要や役割を果たす。エピゲノムは環境に応じて変化することで、環境応答遺伝子の転写されやすさを調節する。特に興味深いのは、栄養素代謝物がエピゲノム修飾の基質として利用される点である。また、エピゲノム制御に関わる酵素も代謝物に依存した活性を示す。これらの代謝物は、栄養摂取、酸素供給、酸化ストレスなどの環境因子の影響下で増減する。つまり、環境に応じたエピゲノムの変化やその結果生じる遺伝子発現や細胞機能の変化は、仲介する代謝物(ハブメタボライト)によって制御される。実際に、さまざまなハブメタボライトが発生、細胞分化、老化、発がん、生活習慣病など、多岐にわたるエピジェネティックな生命現象と深く関わることが報告されている1)。
本稿では、活発に研究が進められてきたハブメタボライトを概観しつつ、最近注目されている新しいエピゲノム修飾や筆者らが取り組んでいるフラビンアデニンジヌクレオチド(FAD)依存性エピゲノム制御機構について解説する。
2.栄養素代謝物によるエピゲノム制御の概要
真核生物の膨大な遺伝情報をコードするゲノムDNAは、ヒストンタンパク質に巻き付けられた状態(クロマチン)でコンパクトに折りたたまれて小さな核内に収納されている。4種類のヒストンタンパク質(H2A、H2B、H3、H4)がそれぞれ2分子ずつ集まり、合計8個のヒストンが複合体を形成する。そこに146塩基対のDNAが巻き付いて、クロマチンの最小構成単位であるヌクレオソームとなる。一般に、転写の起点となるプロモーター領域や転写量の調節を司るエンハンサー領域では、クロマチンは緩んだ状態(オープンクロマチン)になっており、遺伝子発現に必要なRNAポリメラーゼや転写因子がアクセスしやすい状態になっている。
クロマチン構造はエピゲノムと密接に関わっている(図1)。すなわち、DNAやヒストンがアセチル化、メチル化、リン酸化などの修飾を受けることで、ヌクレオソームの化学的性質や他の分子との相互作用に変化が生じ、その結果クロマチンのアクセスしやすさが決まる。一般に、プロモーターやエンハンサーにおいて、ヒストンのリジン残基アセチル化(Kac)やヒストンH3の4番目のリジン残基のメチル化(H3K4me)はオープンクロマチンと強い相関があり、DNAメチル化やヒストンH3の9番目または27番目のリジンメチル化(H3K9me、H3K27me)は転写されにくい凝集クロマチンと結びついている。
エピゲノム修飾には、さまざまな栄養素代謝物が利用される(図2)。例えば、糖や脂肪酸の代謝によって生じるアセチルCoAやアシルCoA は、ヒストンのアセチル化・アシル化の基質となる。また、DNAやヒストンのメチル化にはS-アデノシルメチオニン(SAM)がメチル基供与体として利用されるが、SAM 合成経路はメチオニンや葉酸、コリン、ビタミンB6などの栄養素代謝と連動している。さらに、ケトン体を基質としたヒストンのヒドロキシブチリル化や、後述する乳酸や神経伝達物質によるヒストン修飾など、新たなエピゲノムマーカーが発見され、代謝- エピゲノムネットワークは広がり続けている2)。
エピゲノムの制御には、修飾を導入する酵素(writer)、除去する酵素(eraser)、修飾の有無を認識するタンパク質(reader)が関与する(図1)。これらの役割を担う因子たちが連携することで、エピゲノムの書き換えと維持、転写やクロマチン構造の制御が行われる。これらの中で、eraserは栄養素代謝物に強く依存した活性を示す(図2)。ヒストン脱メチル化酵素は、FADを必須の補因子とするアミンオキシダーゼ型と、α-ケトグルタル酸(αKG)に依存した活性を持つジオキシゲナーゼ型に分けられる。FADはビタミンB2であるリボフラビンに由来する物質であり、αKGはTCA 回路の中間代謝物であることから、両タイプとも栄養摂取や細胞内代謝に強く依存している。また、ヒストン脱アセチル化酵素(HDAC)は、ニコチンアミドジヌクレオチド(NAD+)依存性のsirtuinファミリーとZn2+依存性のHDAC1-11に大別される。NAD+は食事由来のナイアシンやトリプトファン、内因性のニコチンアミドから合成されるため、sirtuinの活性はこれらの物質代謝の影響を強く受ける。HDAC1-11については、短鎖脂肪酸である酪酸によって阻害される。
クロマチン構造は、細胞が置かれた環境または過去に経験した環境によって変化することで、環境への即時応答や長期的な適応を可能にする。上記の代謝物は、栄養、酸素、温度、pHなど、さまざまな環境因子の影響下で動的に制御されているため、環境応答性エピゲノムのハブとして機能することができる。
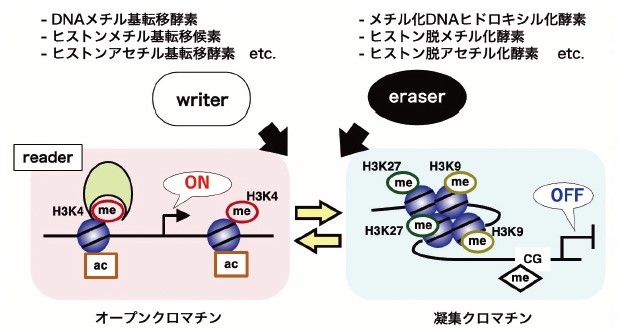
ac: アセチル化、me: メチル化、H3K4,K9,K27: ヒストンH3 の4、9、27番目のリジン残基
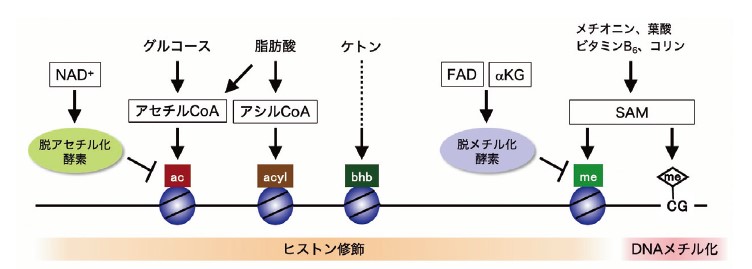
NAD: ニコチンアミドジヌクレオチド、FAD: フラビンアデニンジヌクレオチド、αKG: αケトグルタル酸、CoA: コエンザイムA、SAM: S-アデノシルメチオニン、ac: アセチル化、acyl: アシル化、bhb: βヒドロキシブチリル化、me: メチル化、CG: シトシン- グアニンジヌクレオチド
3.1 乳酸とヒストンラクチル化
乳酸は、近年最も注目されているハブメタボライトの1つである。2019年にヒストンのリジン残基に乳酸が付加されたラクチル化(Kla)の存在が報告されたのを皮切りに、その生物学的役割や制御機構に関する論文が頻繁に見られるようになった(図3)3)。乳酸は解糖の最終産物であり、解糖活性と乳酸産生の間には強い相関がある。そのため、ヒストンラクチル化の研究は、解糖系が著しく活性化されているがん細胞を用いて活発に進められている4)。最近では、神経系や免疫細胞など、さまざまな細胞種を用いた研究が多く行われており、ラクチル化ヒストンはエピゲノムを構成する主要な因子として認識されつつある5)。
Zhangらは、ヒト乳がん細胞やマウスマクロファージ由来のコアヒストンを用いた質量分析により、ヒストンH3K9・K14・K18やH4K5・K8をはじめとして、28箇所のリジン残基がラクチル化されることを明らかにした3)。ヒストンラクチル化は、アセチル化と同様にヒストンの正電荷を減少させることでDNAとの相互作用を弱め、オープンクロマチンを誘導すると予想された。実際に、マクロファージ極性化に伴って発現が上昇する遺伝子の多くでH3K18laレベルの亢進が認められたことから、このマークは転写活性化に関わることが示唆された。その後の多くの研究でも、ヒストンKlaは転写活性化とよく相関することが示されている4)。
ヒストンラクチル化のwriterは、ヒストンアセチル基転移酵素の1つであるp300であることが示されている3)。アセチル化やアシル化の直接の基質がアセチルCoAやアシルCoAであるのと同様に、p300とラクチルCoAの存在下でラクチル化ヒストンが生成されることが実証された。乳酸には光学異性体であるD体とL体が存在するが、解糖によって生じるのはL-乳酸である。培養細胞を用いた実験で、グルコース欠乏や解糖系阻害によりヒストンラクチル化が抑制されたことから、L-乳酸が基質として利用されることが予想された3)。実際に、13CラベルされたL-乳酸から細胞内でL-ラクチルCoAが産生されることが示され6)、また細胞内に内在性のラクチルCoAが検出された7)。さらに、最近ラクチルCoAの合成経路も明らかになってきた。EGFR/ERK経路によってリン酸化されたアセチルCoA合成酵素-2(ACSS2)が核移行し、L-乳酸からL-ラクチルCoAを合成することが報告された8)。別の報告では、脳腫瘍(グリオーマ細胞)でグアノシン三リン酸特異的スクシニルCoA合成酵素(GTPSCS)が核内ラクチルCoA産生を担うことが示された9)。これらの研究により、解糖からヒストンラクチル化までの一連の流れが明らかになった。
一方、ラクチルグルタチオンに由来する酵素に非依存的なヒストンラクチル化経路も存在するが、こちらはD-ラクチルリジンを生み出す。ラクチルグルタチオンもグルコース代謝産物であるメチルグリオキサールに由来するが、D-ラクチル化は細胞内グルコース濃度や解糖フラックスの影響を受けないことが報告されている4)。
11個のZn2+依存性HDACと7個のNAD+依存性HDAC(sirtuin)の系統的な解析により、HDAC3が最も強力なヒストンラクチル化(D-/L-いずれも)のeraserとして働くことが示された10)。Sirtuinの中では、Sirt1、2、および3に脱ラクチル化活性がある。その中で最も活性の強いSirt2は主に細胞質に局在するので、生体内のヒストン脱ラクチル化にはあまり関与しないかもしれない。最近、HDAC3を含むクラスI HDACは、逆にwriterとして働き細胞内の大部分のKla形成に寄与する可能性が報告された11)。興味深いことに、この反応はL-乳酸を基質とするが、ラクチルCoAを介さない。Kla形成にはさまざまなwriter/eraserが複雑に関係しているように思われる。細胞種や基質濃度などさまざまな細胞コンテクストに応じて経路が選択されるのかもしれない。
Zhaiらは、H3K14laレベルが亢進している子宮頸がん細胞において、自作のKlaプローブと結合するタンパク質としてDPF2を同定した12)。DPF2は、ヒストンH3K4のメチル化状態を認識するドメイン構造PHD(plant homeodomain)をタンデムに持つ(double PHD finger)タンパク質で、クロマチンリモデリング複合体の構成因子として転写やエピジェネティックな制御に関わることが知られている。タンパク質構造および機能解析の結果、DPF2のPHDがH3K14laの認識部位であること、ゲノム上でDPF2とH3K14laが高頻度で共存することが明らかになった。DPF2を欠損した子宮頸がん細胞は増殖能の著しい低下を示す。野生型DPF2の導入により回復したが、H3K14laと結合できない変異体では回復しない。これらの結果は、DPF2がH3K14laのreaderとして働くことで、遺伝子制御や細胞機能に寄与することを示唆している。前述のように、H3K14以外にもラクチル化されるリジン残基は多くあるため、他にもKlareaderが存在する可能性がある。
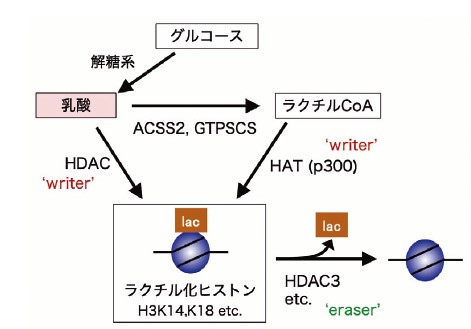
HDAC: ヒストン脱アセチル化酵素、HAT: ヒストンアセチル基転移酵素、ACSS2: アセチルCoA 合成酵素2、GTPSCS: グアノシン三リン酸特異的スクシニルCoA 合成酵素
3.2 モノアミンによるヒストン修飾
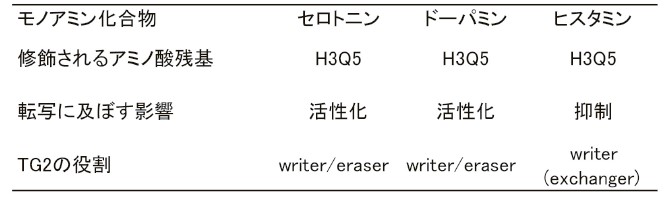
三大栄養素のうち、糖質と脂質の代謝物がエピゲノム修飾の基質として利用される事例が多く報告されている13)。2019年にヒストンのセロトニル化など、アミノ酸代謝物もエピゲノム形成に寄与することが明らかになった14)。セロトニンはトリプトファンの脱カルボキシル化により生成されるモノアミンの一種であり、神経伝達物質として多様な生物学的プロセスに関わる。以前より、transglutaminase-2(TG2)を介してタンパク質内のグルタミン残基にセロトニンを基質としたアミド転移反応(transamidation)が起こることが知られていた15)。マウントサイナイ大学の研究グループは、この反応によりヒストンH3の5番目のグルタミン残基がセロトニル化される(H3Q5ser)ことを示した14)。興味深いことに、H3Q5serは隣にあるH3K4のメチル化を促進し、逆に脱メチル化酵素の反応を抑制することから、転写しやすいクロマチン環境を生み出す「permissive」なマークであることが示唆された。実際に、基本転写因子TFIIDを構成しH3K4me3に結合して転写活性化を起こすTAF3の結合が、H3K4me3/Q5serのdual markにより促進されることが示された。これらの事象から、H3Q5serはH3K4me3を安定化し、遺伝子発現を長期的に維持する役割を持つ可能性が示唆された。さらに、TG2はドーパミンによるヒストン修飾を仲介することも、同じ研究グループにより報告された16)。ドーパミニル基は、セロトニル基と同様にH3Q5に付加され(H3Q5dop)、permissiveなマークとして働くことが示された。H3Qdopは、コカイン中毒を来したラットの症状の長期化(薬物探求行動の持続)に関与する可能性が示された。
同様に、TG2はH3Q5のヒスタミニル化も触媒することが示された(H3Q5his)17)。H3Q5hisはメチル基転移酵素MLL1によるH3K4トリメチル化を抑制することから、ser/dopとは逆に転写抑制性の修飾であることが示唆された。さらに興味深いことに、TG2はserまたはdopが付加されたH3Q5からこれらのマークを除去し、hisを付加する活性を持つことが示された。この機序によるモノアミニル化の交換が遺伝子発現の概日リズムを生み出すことがわかった。この発見は、従来のwriter-reader-eraserパラダイムに、「exchanger」という新しい概念を加える非常に興味深いものである(表1)。エピゲノム制御の奥深さを物語る研究である。
3.3 FAD が仲介する代謝エピゲノムクロストーク
リボフラビン(Rf、ビタミンB2)は、哺乳動物体内では合成できない必須栄養素である。食品添加物やサプリメントとして頻繁に利用され、健康維持に欠かせないビタミンとして広く認識されている。しかし、この古典的な栄養素がどのようにして健康増進効果を発揮するかは明確ではない。特に、長期的な摂取の多寡が私たちの発生過程や老化、疾患発症にどのように作用するかは、知見が乏しい。
体内に取り込まれたRfは各組織に分配された後、細胞内でフラビンモノヌクレオチド(FMN)、FADへと順次変換される。FMNやFADは酸化還元酵素の補因子として、脂肪酸β-酸化やTCA回路など、さまざまな代謝プロセスに関わる。それに加えて、FADはアミンオキシダーゼ型のヒストン脱メチル化酵素LSD1とLSD2の活性にも必須であるため、エピゲノム形成にも直接的な役割を持つ。筆者らは、細胞内FAD合成に依存したLSD1活性化が脂肪細胞の「脂肪貯蔵型」の代謝表現型形成に必須であることを報告し、代謝物によるエピゲノム制御が代謝制御にフィードバックされる「代謝-エピゲノムクロストーク」メカニズムの一端を明らかにした18)。その後の筆者らの研究で、LSD1とLSD2が細胞内外の環境に応じたエピゲノム制御に寄与し、がん細胞固有の代謝や脂肪・肝細胞における脂質代謝、筋肉の質と量の調節に重要な役割を果たすことを明らかにした19-24)。
トリプルネガティブ乳がん(triple negative breast cancer,TNBC)は、エストロゲン受容体、プロゲステロン受容体、および上皮成長因子受容体の発現がいずれも陰性の予後が極めて悪い乳がんサブタイプである。Songらは、ヒトTNBC検体におけるゲノム変異、特に代謝関連遺伝子のコピー数変異を探索した結果、FMNをFADに変換する酵素FADシンセターゼをコードするFLAD1遺伝子が高頻度で増幅されていることを見出した25)。TNBCにおいて、FLAD1のコピー数は発現量と正の相関を示し、FLAD1高発現症例は低発現症例と比較して生存率が有意に低いことがわかった。さらに細胞レベルの解析により、LSD1が転写因子SREBP1(sterol regulatory element-binding protein 1)の発現をH3K9脱メチル化を介して誘導することで、TNBC細胞の生存に必要な脂質合成系遺伝子の発現を促進すること、そのLSD1機能にFLAD1が必須であることが示された。ハブメタボライトとしてのFADの重要性を改めて示す事例である。
FADは、脂肪酸β-酸化酵素の補因子としてアセチルCoA産生に寄与し、葉酸回路を構成する5,10-メチレンテトラヒドロ葉酸還元酵素を介してSAM合成にも関わる。したがって、FAD量の変化はエピゲノム形成に広範な影響を与えると予想される。
4. おわりに:統合的理解に向けて
今世紀初頭からDNA・ヒストン修飾や制御因子が相次いで発見され、その度に調節因子としての栄養素代謝物が注目されてきた。今後も新たなハブメタボライトが数多く発見され、エピゲノムとその制御の多様性がさらに広がっていくであろう。実際には、代謝物によるエピゲノム制御は常に同時多発的であり、各分子機構が相互作用しながらエピゲノムの全体像を構築しているはずである。ビッグデータの解析技術が発達した今日でも、エピゲノム制御の全体像を統合的に理解するための方策はまだ存在しない。今後の重要な課題である。
【参考文献】
- A. Lempradl, J. A. Pospisilik and J. M. Penninger, “Exploring the emerging complexity in transcriptional regulation of energy homeostasis”, Nat. Rev. Genet., 2015, 16(11), 665-681.
- Y. Z. Chen et al., “Association of histone modification with the development of schizophrenia”, Biomed. Pharmacother., 2024, 175, 116747.
- D. Zhang et al., “Metabolic regulation of gene expression by histone lactylation”, Nature, 2019, 574, 575-580.
- M. Iozzo, E. Pardella, E. Giannoni and P. Chiarugi, “The role of protein lactylation: A kaleidoscopic post-translational modification in cancer”, Mol. Cell, 2025, 85, 1263-1279.
- Y. Xie et al., “The role and mechanism of histone lactylation in health and diseases”, Front. Genet., 2022, 13, 949252.
- D. Zhang et al., “Lysine l-lactylation is the dominant lactylation isomer induced by glycolysis”, Nat. Chem. Biol., 2025, 21, 91-99.
- E. L. Varner et al., “Quantification of lactoyl-CoA (lactyl-CoA) by liquid chromatography mass spectrometry in mammalian cells and tissues”, Open Biol., 2020, 10(9), 200187.
- R. Zhu et al., “ACSS2 acts as a lactyl-CoA synthetase and couples KAT2A to function as a lactyltransferase for histone lactylation and tumor immune evasion”, Cell Metab., 2025, 37(2), 361-376.e7.
- R. Liu et al., “Nuclear GTPSCS functions as a lactyl-CoA synthetase to promote histone lactylation and gliomagenesis”, Cell Metab., 2025, 37(2), 377-394.e9.
- C. Moreno-Yruela et al., “Class I histone deacetylases (HDAC1–3) are histone lysine delactylases”, 2025, Sci. Adv., 8(3), eabi6696.
- T. Tsusaka et al., “Class I histone deacetylases catalyze lysine lactylation”, bioRxiv, 2025, doi: 10.1101/2025.02.25.640220.
- G. Zhai et al., “DPF2 reads histone lactylation to drive transcription and tumorigenesis”, Proc. Natl. Acad. Sci. U S A, 2024, 121(50), e2421496121.
- B. R. Sabari, D. Zhang, C. D. Allis and Y. Zhao, “Metabolic regulation of gene expression through histone acylations”, Nat. Rev. Mol. Cell Biol., 2017, 18(2), 90-101.
- L. A. Farrelly et al., “Histone serotonylation is a permissive modification that enhances TFIID binding to H3K4me3”, Nature, 2019, 567(7749), 535-539.
- A. Al-Kachak and I. Maze, “Post-translational modifications of histone proteins by monoamine neurotransmitters”, Curr. Opin. Chem. Biol., 2023, 74, 102302.
- A. E. Lepack et al., “Dopaminylation of histone H3 in ventral tegmental area regulates cocaine seeking”, Science, 2020, 368(6487), 197-201.
- Q. Zheng et al., “Bidirectional histone monoaminylation dynamics regulate neural rhythmicity”, Nature, 2025, 637(8047), 974-982.
- S. Hino et al., “FAD-dependent lysine-specific demethylase-1 regulates cellular energy expenditure”, Nat. Commun., 2012, 3, 758.
- A. Sakamoto et al., “Lysine demethylase LSD1 coordinates glycolytic and mitochondrial metabolism in hepatocellular carcinoma cells”, Cancer Res., 2015, 75, 1445-1456.
- H. Araki et al., “LSD1 defines the fiber type-selective responsiveness to environmental stress in skeletal muscle”, Elife, 2023, 12, e84618.
- K. Kohrogi et al., “LSD1 defines erythroleukemia metabolism by controlling the lineage-specific transcription factors GATA1 and C/EBP α”, Blood Adv., 2021, 5(9), 2305-2318.
- K. Anan et al., “LSD1 mediates metabolic reprogramming by glucocorticoids during myogenic differentiation”, Nucleic Acids Res., 2018, 46(11), 5441-5454.
- K. Nagaoka et al., “Lysine-Specific Demethylase 2 Suppresses Lipid Influx and Metabolism in Hepatic Cells”, Mol. Cell. Biol., 2015, 35(7), 1068-1080.
- R. Takase et al., “Lysine-specific demethylase-2 is distinctively involved in brown and beige adipogenic differentiation”, FASEB J., 2019, 33(4), 5300-5311.
- X. Q. Song et al., “Copy number amplification of FLAD1 promotes the progression of triple-negative breast cancer through lipid metabolism”, Nat. Commun., 2025, 16, 1241.
| [ 著者プロフィール ] | |
| 氏名 | 日野 信次朗(Shinjiro Hino) |
|---|---|
| 所属 |
熊本大学発生医学研究所・細胞医学分野 〒860-0811 熊本県熊本市中央区本荘2-2-1 TEL:096-373-6801 FAX:096-373-6804 |
| 出身学校 | 京都大学農学部 |
| 学位 | 博士(医学)京都大学大学院医学研究科 |
| 専門分野 | 代謝エピジェネティクス |
| 現在の研究テーマ | 栄養素代謝物や内分泌因子によるエピゲノム記憶の形成機序 |