本号より小社が立地する熊本県の生命科学研究最前線を、熊本大学の若手研究者が連載(8回)でお届けします。
動的構造解析による酵素反応機構の解明
Structural dynamics in enzymatic reaction
 |
中村 照也 熊本大学大学院 生命科学研究部 (薬学系) 准教授 |
Abstract
Enzymes are responsible for essential chemical reactions in biological phenomena and catalyze
reactions by binding substrates, coenzymes and metal ions to the active site and reducing the
activation energy. To understand the mechanisms of enzymatic reactions, it is essential not only to
determine the structures of the enzyme-substrate and enzyme-reaction product complexes, but also to
capture the intermediate structures and observe the structural changes during the reaction processes.
X-ray crystallography is a methodology for determining the three-dimensional structure of proteins in
a crystalline state, and in many cases, enzyme activity is retained even in a crystalline state. Here
we present the dynamic structure analysis of enzymes using X-ray crystallography and a new enzymatic
reaction mechanism of MutT, an enzyme that hydrolyzes a mutagenic oxidized nucleotide.
1.はじめに
酵素は、生命現象における様々な化学反応を担うタンパク質であり、基質、補酵素や金属イオンを自身の活性部位に結合し、活性化エネルギーを低下させることで化学反応を触媒する。このような酵素反応機構を理解するには、酵素反応の速度論解析はもちろん、酵素の構造解析が必要不可欠となる。構造解析においては、反応過程における酵素-基質複合体や酵素-反応生成物複合体の立体構造を決定するだけでなく、反応中間体の立体構造も捉えて、反応開始から終了までの構造変化を明らかにすることが求められる。タンパク質構造解析法の一つであるX線結晶構造解析は、結晶状態となったタンパク質の立体構造、すなわち静的なタンパク質の立体構造を決定する手法である。タンパク質の結晶は、溶媒含有率が高く(〜40%以上が溶媒)、結晶状態であっても自身の酵素活性を保持しているケースも少なくない。実際、我々はDNA合成酵素であるDNAポリメラーゼが結晶状態でも酵素活性を保持していることに着目することで、X線結晶構造解析を用いたDNAポリメラーゼの動的構造解析を行った。その結果、DNAポリメラーゼが行うヌクレオチド転移反応において、DNA末端とヌクレオチド(dNTP)の間に共有結合が形成される過程を観察することに成功し、DNAポリメラーゼの新規反応機構を提案した1),2)。さらに近年、突然変異原の酸化ヌクレオチドを加水分解する酵素MutTの動的構造解析も行い、その新規反応機構を提案した3)。本稿では、X線結晶構造解析を用いた酵素の動的構造解析法と、本手法によって最近明らかにしたMutTの酵素反応の新規機構について紹介する。
2.大腸菌MutTタンパク質
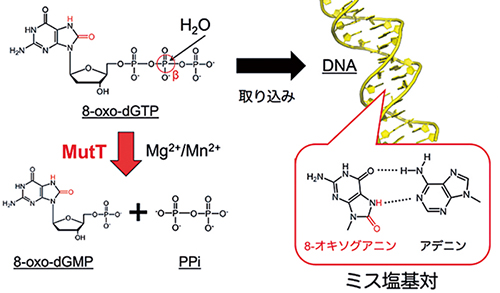
細胞内で生じる活性酸素種は、遺伝情報物質であるDNAだけでなくその前駆体であるヌクレオチドも酸化する。dGTPの酸化により生じた8-oxo-dGTPがDNAポリメラーゼによってDNA中に取り込まれた際、酸化塩基部位の8-オキソグアニンはシトシンだけでなくアデニンともミス塩基対を形成できるため突然変異の原因となる(図1)。1992年、当時九州大学の真木と関口は、本研究対象の大腸菌MutTが8-oxo-dGTPを分解することで8-oxo-dGTPのDNAへの取り込みを防ぎ、突然変異を抑制することを明らかにした4)。これは、遺伝情報エラーの回避がDNAレベルだけでなくヌクレオチドレベルでも行われていることを示した大きな発見であった。
MutTはMg2+もしくはMn2+存在下、8-oxo-dGTPを加水分解する。その際、8-oxo-dGTPのβ-リン原子への水分子の求核攻撃によって、8-oxo-dGTPは一リン酸型の8-oxo-dGMPとピロリン酸(PPi)へと加水分解される(図1)。MutTは、大きな加水分解ファミリーであるNudix/MutTファミリー
(ヌクレオシド二リン酸を加水分解する酵素群)に属し、ファミリー名からもわかる通り、MutTはこのファミリーにおいて最も古くから研究されてきた酵素である。MutTの酵素反応機構は、核磁気共鳴や反応速度論解析などにより研究されており5), 6)、MutTは2つのMg2+/Mn2+イオンを介して加水分解反応を触媒すること(two-metal-ion
mechanism)が提案されていた。一方、これまでに我々が決定したMutTと反応生成物とMn2+の複合体のX線結晶構造(MutT-8-oxo-dGMP-Mn2+)では、MutTが3つのMg2+/Mn2+イオンを介して反応を触媒すること(three-metal-ion
mechanism)が示唆されていた7)。また、他のNudixファミリー酵素においても、加水分解反応の触媒には2つもしくは3つの金属イオンが必要であることが報告されており、統一的な知見は得られていない。これは、Nudixファミリーはわずか23残基という加水分解モチーフのアミノ酸配列で定義されているため、反応機構に多様性がみられると考えることもできる。しかし一方で、酵素反応機構の推定に用いられている結晶構造のほとんどは、基質類縁体や活性を示さない金属イオンなどとの複合体であることや結晶化条件のpHも様々であることにより、酵素反応の活性化状態を反映していない可能性も考えられる。このようなMutTを含めたNudixファミリーの金属イオンを介した酵素反応機構を解明するために、X線結晶構造解析を用いたMutTの動的構造解析を行った。
3.X線結晶構造解析を用いた動的構造解析
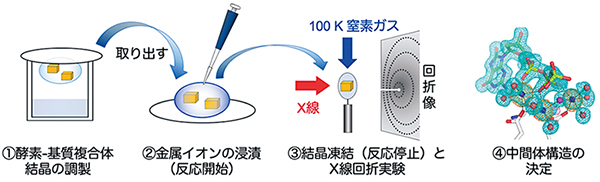
酵素は結晶状態であっても活性を保持しているという特徴を利用して、反応中間体を低温(~100K)で捕捉する低温トラップ法を用いたX線結晶構造解析により、MutTの動的構造解析を行った。MutTやDNAポリメラーゼなど、金属イオン存在下で反応を触媒する酵素については、以下の①〜④の手順で実験を行った(図2)。「①酵素-基質複合体結晶を反応の起こらない状態で調製する。②反応開始となる金属イオンを結晶に浸漬させて結晶内反応を開始させる。③結晶を目的の反応時間インキュベートした後、急速凍結して反応を完全に停止させる。④反応時間毎の中間体構造をX線結晶構造解析により決定する。」という手順である。本動的構造解析を行うにあたっては、1)反応の起こらない状態で酵素-基質複合体構造 (ES、反応開始前構造) を決定できる、2)追跡可能なタイムスケールで反応を進行できる、3)反応中に結晶の状態を維持できる(結晶が崩壊するとX線回折データを収集できないため)、などといった条件を満たす必要があるため、結晶化条件のみならず、金属イオン濃度、pH、反応時間、反応温度といった条件を検討し、反応過程を追跡できる結晶内反応条件を酵素に応じて見出す必要がある。実際、DNAポリメラーゼとMutTについては、それぞれ100以上の結晶内反応条件を検討することで、最終的に反応開始から終了までの全過程を追跡することが可能となった1), 3)。DNAポリメラーゼの場合は、pH7.0で反応時間が40~300秒で酵素反応過程を観察できたが、MutTの場合は、pHを5.8にして反応速度を下げ、反応時間を数十分から数時間とすることで反応過程を観察することができた。
4.酵素反応過程の観察と新規反応機構
MutTの動的構造解析により、ES(MutT-8-oxo-dGTP複合体)、EP(MutT-8-oxo-dGMP複合体)を含む合計6つの中間体構造を決定し、MutTによる8-oxo-dGTPの加水分解反応過程を原子レベルで観察した(図3)。なお本稿では、これらのうち4つの構造を示している。反応開始前のESでは、8-oxo-dGTPの三リン酸部位がMutT活性部位のGly37、Glu57、Glu53に水分子とNa+イオンを介して結合しているが(図3のES)、三リン酸部位では、活性部位に近づいている配座Aと活性部位から遠ざかった配座Bの2つの状態がみられた。これは、酵素活性に必須のMg2+/Mn2+イオンのない時は、三リン酸部位が活性部位に引きつけられない、すなわち、活性状態にはないことを示唆している。結晶をMn2+溶液に浸漬させることで結晶内反応を開始した。反応の開始と共に、まず1つのMn2+(Mn2+A)がGly37、Glu57、三リン酸に配位し、このMn2+Aの配位によって三リン酸部位は活性部位に引きつけられる(図3のState1の矢印)。反応時間が経過すると、さらに2つのMn2+(Mn2+BとMn2+C)が活性部位に配位する(図3のState2)。これら3つのMn2+が活性部位に配位した後、Mn2+BとMn2+Cに架橋する水分子が現れる。このように、MutTの活性部位に3つのMn2+が配位することで、8-oxo-dGTPの三リン酸部位が活性部位に引きつけられるとともに、水分子が8-oxo-dGTPのβ-リン原子を求核攻撃するために適した位置に配置される。この水分子がβ-リン原子を求核攻撃することで、8-oxo-dGTPが8-oxo-dGMPへと加水分解されることが明らかとなった。反応時間が経過すると、8-oxo-dGTPは8-oxo-dGMPへと加水分解され、PPiとMn2+BとMn2+Cは活性部位から遊離する(図3のEP)。また、構造解析での電子密度図から、State2で現れた水分子は、ESとState1においてGlu53と結合していた水分子が、State2にてMn2+BとMn2+Cに架橋するように移動したことが示唆された。過去の反応速度論解析において、このGlu53が反応における一般塩基となることが報告されていることから6)、現段階では、図3の下段に示したようにGlu53がMn2+の配位子として、さらには一般塩基として働くという酵素反応機構を提案している。また、Mg2+を用いた酵素反応過程の追跡も行ったが、Mn2+の場合と同様、MutTは3つのMg2+を介して反応を触媒することが明らかとなった。
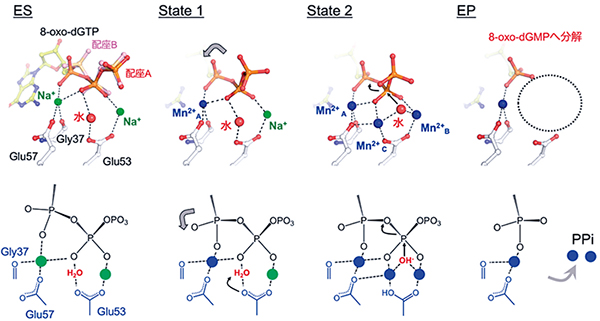
5.おわりに
酵素の動的構造解析により、反応開始から終了までの酵素反応過程を観察することで、MutTが3つの金属イオンを介して8-oxo-dGTPを加水分解すること(three-metal-ion mechanism)を解明し、新規反応機構を提案することができた。本研究により、Nudixファミリーの中にはthree-metal-ion mechanismで加水分解反応を触媒する酵素も存在することが示唆された。図3の下段に示した酵素反応機構では、求核攻撃を行う水分子の脱プロトン化も含め、水素原子を含んだ酵素反応機構を議論している。しかしながら、X線結晶構造解析では水素原子を観察することが極めて難しいため、提案した反応機構における水素原子の位置と動きは、今回の動的構造解析の結果とこれまでの反応速度論解析の結果から推測している。反応におけるプロトン化/脱プロトン化を含めた水素原子の位置および動きを明らかにすることは、タンパク質の機能を原子レベルで議論する上で極めて重要であるが、タンパク質構造解析の現状としては、多くの場合、水素原子の位置は推測するにとどまっている。今後は水素原子を高感度で検出できる中性子構造解析も組み合わせることで、反応機構を水素原子レベルで議論できるような酵素反応の物理化学的研究を展開したいと考えている。
6.おわりに
20年前から考えると今のプロテオミクスは夢のようである。プロテオミクスのアプローチが多く、網羅性が高い方法もあれば1個の細胞からでもプロテオミクス情報を取得できる高感度な方法もある。このことを考えると、今のプロテオミクスにできないことでも数年後にはできるようになっている可能性は高く、プロテオミクスは生命科学研究にますます貢献できる分析技術となるだろう。本稿が生命科学研究者にとってプロテオミクスを積極的に利用していただくきっかけになることを期待している。
| [ 著者プロフィール ] | |
| 氏名 | 中村 照也 (NAKAMURA Teruya) |
|---|---|
| 所属 | 熊本大学大学院生命科学研究部 (薬学系) 〒862-0973 熊本市中央区大江本町5-1 TEL : 096-371-4638 |
| 出身学校 | 大阪大学大学院薬学研究科博士前期課程 |
| 学位 | 博士 (薬学) (熊本大学) |
| 専門分野 | 構造生物学 |
| 現在の研究テーマ | ゲノム安定性、細胞シグナル伝達に関わる構造生物学 |