本号より小社が立地する熊本県の生命科学研究最前線を、熊本大学の若手研究者が連載(8回)でお届けします。
次世代プロテオミクス技術の開発と展開
Development and application of next-generation proteomics technologies
 増田 豪 慶應義塾大学大学院
政策・メディア研究科 先端生命科学研究所 特任講師 |
 大槻 純男 熊本大学大学院
生命科学研究部 教授 |
Abstract
Proteomics is a method for comprehensively quantifying proteins present in a sample. In the past,
proteomics technologies had low protein coverage and sensitivity. In recent years, the development of
related technologies has been tremendous, and it is now possible to quantify 10,000 different proteins
in a single shot of liquid chromatography tandem mass spectrometry (LC-MS/MS) by using the developed
MS method and data analysis software. In addition, it possible to obtained proteomic information even
from a single cell by developed sample preparation methods. Recent developments enable to observe not
only protein-protein interactions but also protein-small molecule interactions. Protein observation is
indispensable for life science research. In this article, we hope researchers will find that
proteomics a useful technology and apply the proteomic techniques into their own research to help
clarify their questions.
1.はじめに
タンパク質は生命活動にとって重要な分子群である。タンパク質は単独で機能することは少なく、タンパク質や代謝物、DNAなどその他生命分子と相互作用することで特有な機能を発動する。また、細胞内外で発生したシグナルを伝達するためにタンパク質群が複雑なネットワークを形成している。プロテオミクスは試料中に存在するタンパク質を網羅的に観察する方法であり、タンパク質の役割を明らかにするために生命科学分野で多く用いられている。
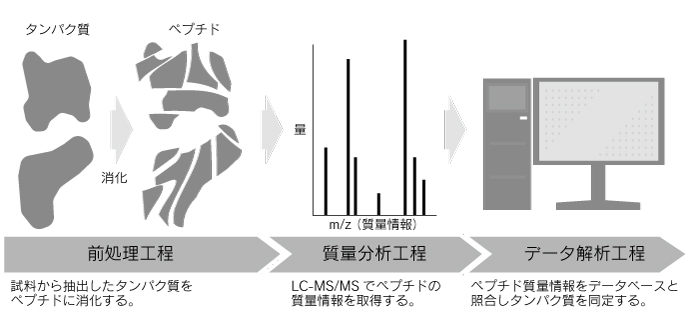
プロテオミクスは前処理、質量分析およびデータ解析の3工程に大きく分かれている(図1)。前処理工程では、試料からタンパク質を抽出し消化酵素でペプチドにする。質量分析工程では、前処理工程で得られたペプチドを液体クロマトグラフィータンデム質量分析(LC-MS/MS)に導入し、ペプチドの配列を質量情報として取得していく。データ解析工程では、分析工程で得られた質量情報からソフトウェアを用いてペプチドおよびタンパク質の定性および定量を行う。これら各工程に用いられる手法は年々改良・開発され、20年前に比べてプロテオミクスの用途は大きく広がった。特に網羅性や高感度性能は飛躍的に向上している。本稿では近年のプロテオミクスの発展に寄与したこれら2つの技術について主に紹介する。
2.網羅性の改善
ヒトの遺伝子は約20000種類あるとされ、そのうち約1万数千種類がタンパク質として発現していると考えられている1)。プロテオミクスはかつて、同定できるタンパク質の網羅性の低さが課題だった。以前は質量分析計でペプチドを測定する場合、data
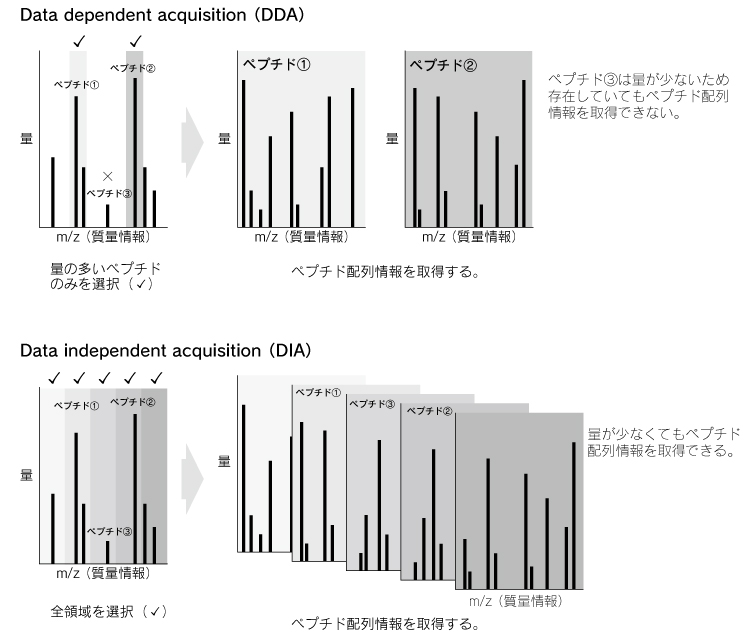
dependent acquisition (DDA)でペプチドの配列情報を取得することが一般的だった。DDAは、検出量が多い順にペプチドを選択し、それらについてペプチド配列情報を取得する方法である(図2上段)。LC-MS/MSでは一度に複数のペプチドが同時に検出されるため、質量分析計の処理スピードが追い付かない場合は配列情報を取得できないペプチドが出てくるため同定結果の再現性が低かった。そこでこれまで、タンパク質やペプチドをその生化学的特性にしたがって分画2) 3)したりLCの分離能を上げる4)ことで問題を解決しようとしてきた。これらの方法を用いることで大幅に同定数は増加したものの、それでもプロテオミクス結果の低い再現性は解決できなかった。
現在はdata
independent acquisition
(DIA)で測定することでこの問題を解決できている。DIAはペプチドの検出量情報に依存せず、質量分析計に導入された全てのイオンについて配列情報を取得する方法である(図2下段)。そのため、DIAはDDAモードに比べてデータ間で同定されるタンパク質の再現性が高くなる。一方で、DIAでは正しくペプチドを同定するためにDDAデータと照らし合わせる必要があったため、同定できるタンパク質の数はDDAデータに依存していた。近年、ニューラルネットワークなどを用いて過去の質量分析データを学習させることで、アミノ酸配列からペプチド配列情報を高精度に予測できるようになった5)。このことから、DDAデータが不要となりFASTAファイルでDIAデータの正確な解析が可能となった。LC-MSの高度化も合わさり、DIAで測定することで1回の分析で10000種類を超えるタンパク質を定量できることが報告されている6)。
DIAは現在、プロテオミクスの一般的な質量分析方法となった。一方で翻訳後修飾解析やマルチプレックス解析などDDAが得意とする部分もあり、使い分けることで目的に即したデータを取得することが求められる。
3.高感度化
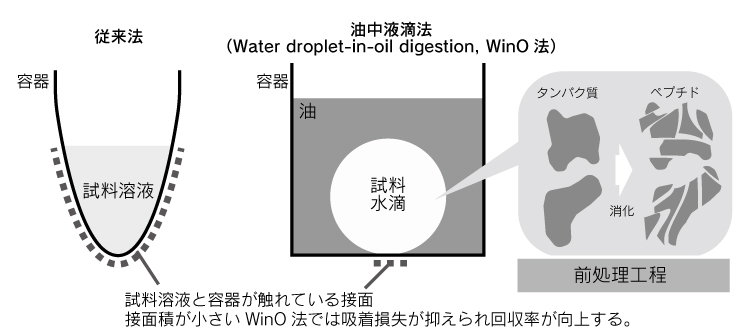
一般的なプロテオミクスでは200~1000ngの消化したペプチドをLC-MS/MSに導入する。これだけの量のペプチドを調製するには初期試料として1万個程度の細胞が必要である。一方で、疾患組織片やセルソートした希少細胞集団など、1万個の細胞に満たない微量試料についてもプロテオミクスを行いたいといった需要も多い。微量試料を高感度に分析するために、これまでLC部分を中心に改良されてきた7)。近年では、1細胞プロテオミクスへの展開が期待され、さらなる高感度分析が必要とされてきた。質量分析計自体は1細胞に含まれるほとんどのタンパク質を検出できるほど十分に高感度であることが知られている8)。一般的な前処理方法で1細胞のプロテオミクスを行うとタンパク質はほぼ同定できないことから、現在は前処理方法の高感度化が行われている。1細胞プロテオミクスにおける一般的な前処理方法の問題点は、1細胞から抽出した極微量のタンパク質が吸着損失してしまうことである(図3)。タンパク質は遺伝子のようにそれを鋳型に増幅することができないため、いかに損失することなくLC-MS/MSに導入するかが重要となる。1細胞プロテオミクスを可能とするために前処理方法がこれまでいくつか報告されている9) 10)。1細胞プロテオミクスの前処理技術には高回収率でありマルチウェルプレートを用いて1度に複数の1細胞を同時に処理できることが求められる。さらに1細胞プロテオミクス研究領域の間口を広げるためには誰もが使用できるような簡便な手法が必要であり、開発した研究チームしか持っていないような特殊な装置を使わない技術が望ましいと筆者は考えている。筆者らはこれら要件を満たす前処理技術として油中液滴法(Water droplet-in-oil digestion, WinO)を開発した(図3)11)。WinO法は油の中に形成した微小な水滴でプロテオミクスの前処理を行う方法である。WinO法では、試料溶液の形状が球体に近づきチューブとの接面積が大幅に縮小し回収率が従来の消化方法に比べて10倍向上した。セルソーターで細胞をマルチウェルプレート中の油中液的に直接導入でき、液体ハンドリングロボットで前処理を行える。また、質量分析計で検出されたペプチドイオンの由来細胞を見分けられるタグとなる安定同位体標識試薬を用いることで最大16個の1細胞を混合して1回のLC-MS/MSでデータを取得できる。1個の細胞から約800種類のタンパク質を定量できるWinO法は1細胞プロテオミクスに有用な簡便な方法である。
4.その他のプロテオミクス技術とその利用
上述した技術以外にも、プロテオミクスはいろいろな前処理方法と組み合わせることで様々な用途に利用されてきている。例えば、抗体などを用いたアフィニティーカラムにより標的を濃縮して、翻訳後修飾情報や相互作用情報を取得することができる。相互作用解析においては近年、近接依存的標識できる酵素を用いた方法が利用されている。標的タンパク質にTurboID12)やAPEX213)などの近接依存的標識酵素をフュージョンさせて発現させることで、弱いもしくは短時間の相互作用タンパク質でも同定できるようになった。また、この方法は細胞内の標的タンパク質が局在している部位で標識が完了するため、標識後の試料調製時に相互作用してしまう偽陽性タンパク質を減らすことができることが大きな利点である。タンパク質-タンパク質相互作用以外にも、サーマルシフトアッセイと組み合わせることで、低分子化合物と相互作用するタンパク質候補を網羅的に同定することができる14)。サーマルシフトプロテオームプロファイリングは、低分子と相互作用するタンパク質の熱安定性が上昇することを利用して、熱変性温度が上昇したタンパク質を網羅的に探索する手法である。この方法は培養細胞、血清や組織など幅広く応用可能されており、またサーマルシフトアッセイ方法を改良することで膜タンパク質と低分子化合物の相互作用解析にも用いられている15) 16)。
5.今後の展開
プロテオミクスの今後期待される展開として網羅的なプロテオフォーム解析手法の確立がある。同じ遺伝子から発現したとしても異なるプロセッシングを受けた多様なプロテオフォームが細胞内には存在する。プロテオフォームはタンパク質の機能を理解するうえで重要であるものの、プロテオミクスではペプチドまで断片化するためプロテオフォーム情報は失われてしまう。プロテオフォームを網羅的に解析するために、長いペプチドやタンパク質を測定できる前処理方法、質量分析方法やデータ解析手法の開発が重要になってくるだろう。また、大規模なプロテオームデータから生命現象を説明するために必要な解析手法の開発も今後期待される課題の一つである。遺伝子のノックアウトや薬剤投与など細胞に摂動を与えると多くのタンパク質の発現量が変動する。大規模なプロテオミクスデータから得られる数多くの変動タンパク質からどれを選ぶべきなのか迷ってしまうことは多々ある。プロテオミクスを行えば発生している生命現象を簡単に捉えられるわけではない。研究課題に沿ったタンパク質を候補群の中から絞り込む解析手段を充実させることでプロテオミクスデータの有用性がさらに向上すると期待している。
6.おわりに
20年前から考えると今のプロテオミクスは夢のようである。プロテオミクスのアプローチが多く、網羅性が高い方法もあれば1個の細胞からでもプロテオミクス情報を取得できる高感度な方法もある。このことを考えると、今のプロテオミクスにできないことでも数年後にはできるようになっている可能性は高く、プロテオミクスは生命科学研究にますます貢献できる分析技術となるだろう。本稿が生命科学研究者にとってプロテオミクスを積極的に利用していただくきっかけになることを期待している。
| [ 著者プロフィール ] | |
| 氏名 | 増田 豪(Takeshi Masuda) |
|---|---|
| 所属 |
慶應義塾大学大学院政策・メディア研究科/先端生命科学研究所 〒997-0017 山形県鶴岡市大宝寺日本国403-1 TEL: 0235-29-0572 FAX: 0235-29-0536 |
| 出身学校 | 東京農業大学 |
| 学位 | 博士(生物産業学) |
| 専門分野 | プロテオミクス、分析化学 |
| 現在の研究テーマ | 1細胞プロテオミクス技術の開発とその利用、多発性骨髄腫の薬剤耐性機構解明 |
| 氏名 | 大槻 純男(Sumio Ohtsuki) |
|---|---|
| 所属 |
熊本大学大学院生命科学研究部 〒862-0973 熊本県熊本市中央区大江本町5-1 TEL: 096-371-4323 FAX: 096-371-4329 |
| 出身学校 | 東京大学 |
| 学位 | 博士(薬学) |
| 専門分野 | プロテオミクス、生物薬剤学 |
| 現在の研究テーマ | DIAプロテオミクス開発と血液脳関門の機能解明 |