 |
| トップページ > 一分子DNAポリメラーゼによるリアルタイムDNA シーケンシング |
 |
一分子DNAポリメラーゼによるリアルタイムDNAシーケンシング株式会社 同仁化学研究所 江頭貴宏 今日、ゲノム情報の解明が盛んに行われている。なかでも、ヒ トのゲノムの全塩基配列を解析するプロジェクトであるヒトゲノ ム計画(Human Genome Project)が、2003年に完了し、遺伝 子疾患の原因追究やその治療法開発だけでなく、ヒトの進化の歴 史の解明など、バイオ技術、生命科学に応用できることが期待さ れている。 ゲノムの塩基配列を解析する手法としては、サンガー法やそ の改良法があり、これらはDNAポリメラーゼを利用したもので ある。サンガー法では低濃度の鎖停止ヌクレオチド(ターミネー ターと呼ばれる)などを使って読み取りを中断し、電気泳動など で分離して解析を行っている。従ってこの方法は、操作が煩雑で、 解析に時間がかかってしまう(図1)。 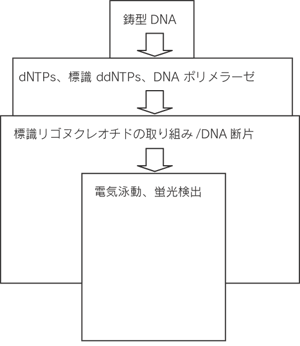 図1
その方法は、一分子のDNAポリメラーゼを使用し、DNA合 成中に取り込まれた蛍光標識ヌクレオチドをリアルタイムで検 出する方法1) や、ヌクレオチドがDNA合成中に取り込まれる時 に放出するピロリン酸をATPに変換し発光反応に用いることで、 DNA合成中に使われたヌクレオチドを定量するピロリン酸塩検 出法塩基延長法がある2)。また、化学的な識別とは全く異なる、 ヌクレオチドの各塩基を物理的に識別することによって配列を決 定するナノポア法がある3)。ナノポア法は半導体技術を応用した もので、一本鎖DNAを微小なSi酸化膜を通過させ、その時に流 れる電流を測定し塩基配列を解読する。この方法は操作が簡単で 安価であること、しかも、蛍光色素などの試薬を必要としないと いう大きなメリットを持つが、一分子の塩基を識別出来る解像度 が得られるかどうかが課題である。 ここでは、次世代の塩基配列解析法の中で、特に一分子のDNAポリメラーゼを固定化し、リアルタイムでDNA合成を観察 する方法を紹介する。 この方法はPacifi c Bioscience社が開発し、短時間でDNA配 列を読み取ることができ、読み取れる鎖長が従来法よりも長く、 ゲノムの解析の速度を大幅に増やすことができるようになると期 待されている(図2)。 この方法の特徴は、それぞれ違う波長をもつ蛍光色素を塩基で はなく末端のリン酸につけた4種類のデオキシリボヌクレオシド 三リン酸(dNTPs)(図3)を使うことである。また、Zero-Mode Waveguide(ZMW)を開発し、鋳型DNAに従って連続的に合成を行う一分子のDNAポリメラーゼが、dNTPsを取り込む間の蛍光 のみ検出できるようになり、リアルタイムでDNA塩基配列の決 定に成功した。 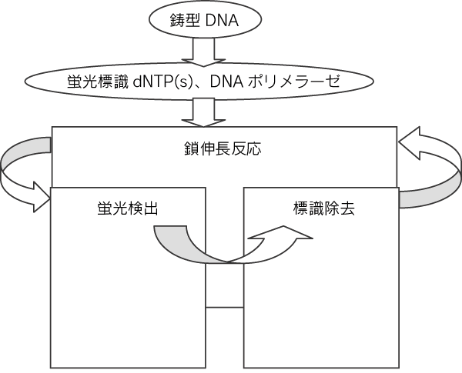
図2 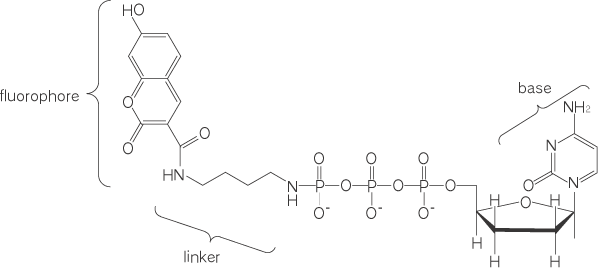
図3 リアルタイムで複数のDNAポリメラーゼのDNA合成を一度 に検出すると、各々のDNAポリメラーゼの合成速度にバラツキ があるため正確な測定ができない。そのため、DNA合成を個別 に観察する必要がある。そこで、一分子のDNAポリメラーゼを 直径100nmのガラス基盤に独立に吸着させ、DNA合成の様子を 個別に観察した。 DNAポリメラーゼを吸着させるガラス底面には、ビオチン化 したポリエチレン・グリコールを結合させ、DNAポリメラーゼ1分子のみを固定化した。また、側面への非特異吸着を抑えるた めに、側面のアルミニウム表面をポリリン酸で修飾している(図4)。 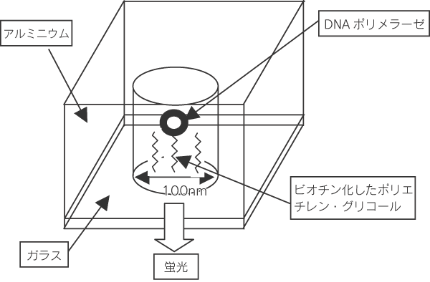
図4 DNAポリメラーゼによるDNA合成を蛍光検出する場合は、蛍 光ラベルを持つヌクレオチドが必要になる。共焦点顕微鏡や全反 射顕微鏡法などの照射範囲が広いものを使うとDNA合成の活性 部位に取り込まれていない蛍光ラベルを持つヌクレオチドの蛍光 が観測されてしまいバックグラウンドが高くなるという問題点が ある。そのため、ZMWを使って狭い範囲を照射し、DNAポリメ ラーゼ一分子にしか光が届かないように設計されている。 リアルタイムでのDNA配列の決定は以下のようにして行わ れる(図5)。鋳型一本鎖DNAの塩基に相補的な蛍光ラベルしたdNTPがDNAポリメラーゼにより取り込まれ、蛍光基もZMWの検出範囲内に入ってきて蛍光が検出される。DNA合成が終了 すると、蛍光ラベルしたリン酸ジエステル結合が外れて拡散し、 より蛍光基がZMWの検出範囲外に出ていくため、蛍光が検出さ れなくなる。次に、別の蛍光ラベルしたDNAの塩基に相補的なdNTPがDNAポリメラーゼにより取り込まれ、DNAが合成され る間、蛍光が検出され、DNA合成が終了し、蛍光ラベルしたリ ン酸ジエステル結合が外れると蛍光が検出されなくなる。この検 出を続けて、検出された蛍光波長の種類と順番により、DNAの 配列が決定される。 図4に示した一分子のDNAポリメラーゼを固定したZMWを、 数千を集積して1枚のチップにすることが考えられている。この チップを用いると、数千の鋳型DNAのDNA配列を一度に観測 でき、多くのDNA解析ができる。 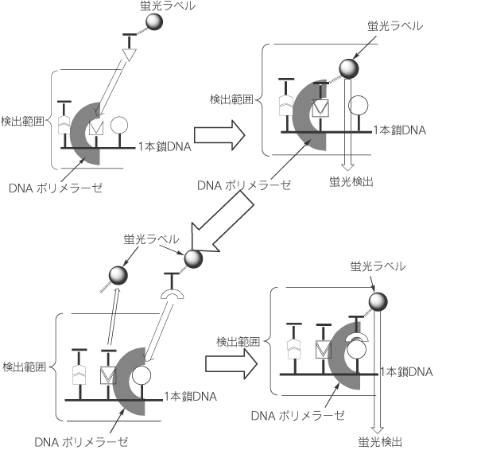
図5 ここで紹介したDNAシーケンシング方法を使うことで、解析 スピードが増し、さらに一度に多種類のDNAを分析することで コストの低減にも繋がる。また、多くの解析データを蓄積するこ とができるようになる。その結果、ヒトの遺伝子と染色体の根本 的な構成がより明らかになり、一人一人にあわせたテイラーメイ ド医療を行なえるなど、治療や予防が画期的に進展することが期 待されている。 参考文献
1) J. Eid, et al., Science, 2009, 323 , 133-138
|
| Copyright(c) 1996-2010 DOJINDO LABORATORIES,ALL Rights Reserved. |